
Comparaison entre une méthode de cartographie fine et des tests d'association dans le cadre de l'étude d'un caractère génétique avec des données incomplètes
Mots-clés : SNP, cartographie génétique, processus de coalescence, ARG, fonction de pénétrance, imputation, test d'association.
Ce mémoire porte sur des études d'association entre des variations sur le génome et un trait d'intérêt (maladie, par exemple). Dans une telle étude génétique, il arrive souvent que plusieurs génotypes de certains individus soient manquants dans un échantillon en raison d'un défaut expérimental. Pour pouvoir trouver le lien entre des variations· génétiques et ce caractère d'intérêt avec ce genre de données, on choisit en général une méthode d'imputation permettant d'inférer les génotypes manquants afin d'appliquer un test d'association. Cette méthode utilise un panel de référence qui contient des génotypes provenant de quelques individus sélectionnés dans une population type, génotypes qui se situent dans la même région du génome que les génotypes manquants. Malgré que le test d'association (SNPTEST) analyse des données imputées en prenant en considération l'incertitude de l'imputation, l'information obtenue à travers le panel de référence est limitée et cela peut causer une imputation inexacte, ce qui a un impact sur le test d'association. Une réponse à ce type de problème est d'utiliser une méthode qui ne demande pas de compléter les données manquantes afin de faire les analyses: DMap. C'est une méthode de cartographie génétique fine qui s'appuie sur la théorie de la coalescence et construit des généalogies des individus de l'échantillon sans nécessairement connaître tous les génotypes. L'objectif de ce mémoire est de décrire et d'évaluer ces deux méthodes qui servent à analyser les données génétiques incomplètes dans un échantillon. Nous voulons surtout comparer la performance de la méthode DMap avec celle de la méthode SNPTEST en testant leur sensibilité à plusieurs paramètres, dont la fréquence de la maladie dans la population et la proportion de données manquantes dans l'échantillon. Ce mémoire est divisé en 6 chapitres. Le premier chapitre contient une introduction à quelques concepts de base en génétique qui seront utilisés dans la suite. Le deuxième chapitre présente la théorie de la coalescence, tandis que la méthode DMap est présentée en détails au troisième chapitre. Au quatrième chapitre, nous décrivons la méthode IMPUTE qui permet d'inférer les génotypes manquants dans un échantillon. Par la suite, la méthode SNPTEST est présentée au cinquième chapitre. Nous ter~inons ce mémoire, avec le sixième chapitre, qui présente une étude de simulation afin de comparer les résultats obtenus par les deux méthodes, DMap et SNPTEST.
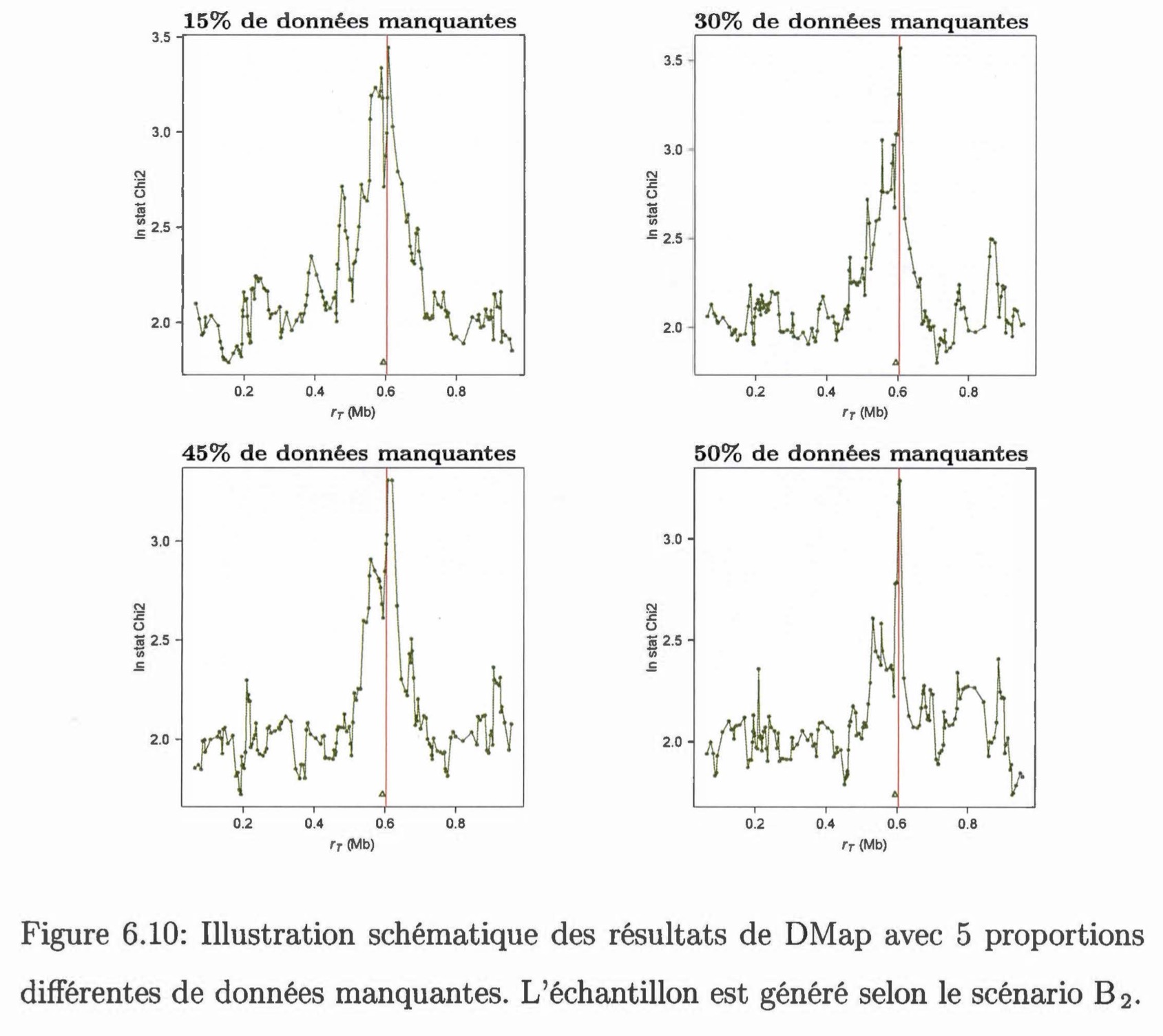
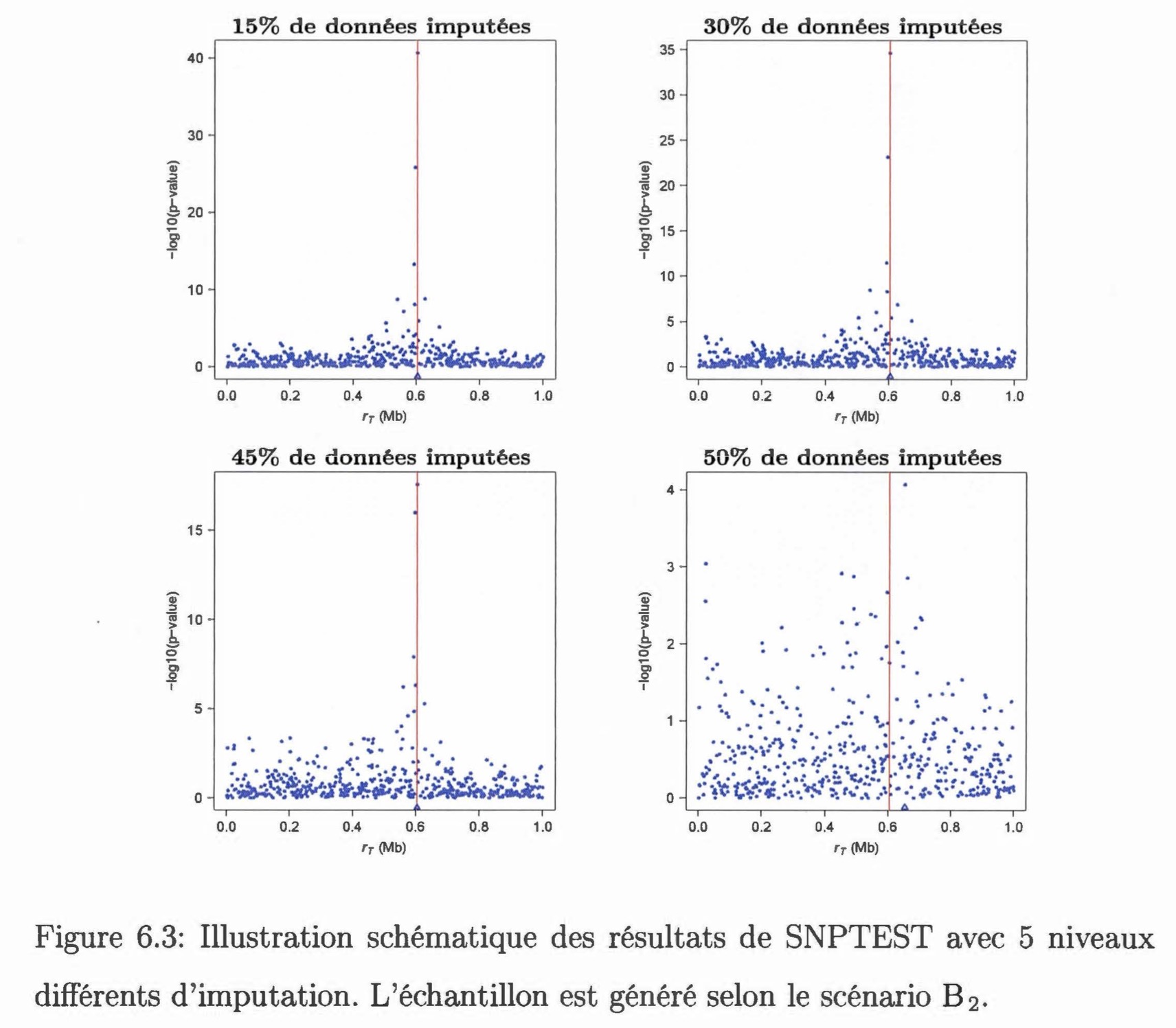
L'objectif de ce mémoire était de comparer l'efficacité de deux méthodes permettant de tester le lien entre un caractère d'intérêt et des marqueurs génétiques avec des données incomplètes. La première méthode, DMap, analyse directement les données incomplètes ; cette méthode suppose que les haplotypes sont connus et tient compte de la dépendance ancestrale entre les individus de l'échantillon. De ce fait, DMap peut se dispenser de l'imputation des données. La deuxième méthode, SNPTEST, effectue des analyses en faisant appel à l'imputation des données et prend en considération l'incertitude d'imputation. Nous avons évalué et comparé la performance de ces deux méthodes par une étude de simulation. Sept scénarios ont été proposés pour générer différents échantillons, et ces scénarios varient par rapport au risque relatif et à la fréquence de la maladie. Pour chaque scénario, 5 proportions ont été choisies afin de simuler des données manquantes : on considère que 5%, 15%, 30%, 45% et 50% de génotypes sont manquants. D'après les résultats obtenus, les deux méthodes ont montré une sensibilité au risque relatif et à la fréquence de la maladie. Malgré cela, les deux méthodes semblent très efficaces quand il n'y a pas plus de 45% de données manquantes dans l'échantillon. Lorsqu'il y a une moitié de données manquantes dans l'échantillon, la méthode SNPTEST a de la difficulté à donner une bonne estimation de la position du TIV, tandis que la méthode DMap réussit à trouver la vraie position du TIV si l'on augmente le nombre de marqueurs à analyser. Ainsi, si la fonction de pénétrance est connue, il serait préférable d'utiliser la méthode DMap quand la proportion de données manquantes atteint 50%.
